El Laboratorio de Metagenómica y Metatranscriptómica forma parte del Centro Regional de Estudios Genómicos (CREG), Facultad de Ciencias Exactas de la Universidad Nacional de La Plata (UNLP), y se integra como parte integrante al CENEXA (Centro de Endocrinología Experimental y Aplicada, CONICET-UNLP).

La Dra. Christina B. McCarthy dirige las líneas de investigación que se desarrollan en el laboratorio.
En este laboratorio se analiza el microbioma de diversos y diferentes ambientes para descubrir cómo utilizarlo para beneficio humano. El microbioma se puede definir como la colección de microorganismos y condiciones de un ambiente particular, y hay un número creciente de estudios que lo vinculan con la salud humana y la sustentabilidad ambiental, entre otros. La metagenómica permite caracterizar e inferir la función potencial del microbioma, mientras que la metatranscriptómica permite obtener una imagen de su perfil funcional (y taxonómico) activo. Actualmente se analiza el metatranscriptoma asociado a diferentes insectos (vectores de enfermedades infecciosas e insectos plaga), y el metagenoma asociado a suelos prístinos. Los campos de aplicación incluyen la búsqueda de potenciales agentes de control biológico y la biorremediación de suelos contaminados, entre otros.
Líneas de Investigación
A continuación, se presentan las principales líneas de trabajo que se desarrollan actualmente en el laboratorio:
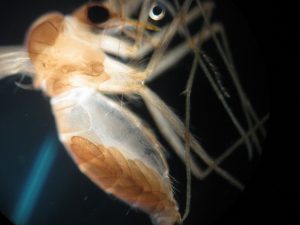 La microbiota tiene roles importantes y muchas veces esenciales durante el crecimiento y desarrollo de muchas especies de insectos. Por otro lado, los microorganismos asociados a vectores de diferentes enfermedades podrían ejercer un efecto patogénico directo sobre el hospedador, interfiriendo con su reproducción o reduciendo su competencia vectorial. Sin embargo, a pesar de su importancia, todavía es escasa la información disponible para muchas especies de insectos. En este contexto, trabajamos con dos insectos: Lutzomyia longipalpis, insecto flebótomo vector de leishmaniasis visceral, y Spodoptera frugiperda, lepidóptero plaga del maíz.
La microbiota tiene roles importantes y muchas veces esenciales durante el crecimiento y desarrollo de muchas especies de insectos. Por otro lado, los microorganismos asociados a vectores de diferentes enfermedades podrían ejercer un efecto patogénico directo sobre el hospedador, interfiriendo con su reproducción o reduciendo su competencia vectorial. Sin embargo, a pesar de su importancia, todavía es escasa la información disponible para muchas especies de insectos. En este contexto, trabajamos con dos insectos: Lutzomyia longipalpis, insecto flebótomo vector de leishmaniasis visceral, y Spodoptera frugiperda, lepidóptero plaga del maíz.
 La biorremediación involucra el uso de microorganismos autóctonos o introducidos (alóctonos) para degradar, transformar y/o esencialmente eliminar contaminantes del suelo y agua. Los extremófilos son microorganismos adaptados a vivir en condiciones extremas físicas y/o geoquímicas, que han desarrollado extremozimas con un gran potencial biotecnológico. El enfoque metagenómico genera un valioso banco de datos donde encontrar nuevos genes que codifican productos génicos biotecnológicamente útiles. En las últimas décadas la agricultura argentina, de producción intensiva, está dejando innegables secuelas de erosión y susceptibilidad a la socavación, además del empobrecimiento de la biodiversidad y de los ecosistemas. En función de generar herramientas con potencial para la biorremediación de suelos, se está caracterizando la composición de microorganismos capaces de proliferar en ambientes extremos y sus extremozimas asociadas.
La biorremediación involucra el uso de microorganismos autóctonos o introducidos (alóctonos) para degradar, transformar y/o esencialmente eliminar contaminantes del suelo y agua. Los extremófilos son microorganismos adaptados a vivir en condiciones extremas físicas y/o geoquímicas, que han desarrollado extremozimas con un gran potencial biotecnológico. El enfoque metagenómico genera un valioso banco de datos donde encontrar nuevos genes que codifican productos génicos biotecnológicamente útiles. En las últimas décadas la agricultura argentina, de producción intensiva, está dejando innegables secuelas de erosión y susceptibilidad a la socavación, además del empobrecimiento de la biodiversidad y de los ecosistemas. En función de generar herramientas con potencial para la biorremediación de suelos, se está caracterizando la composición de microorganismos capaces de proliferar en ambientes extremos y sus extremozimas asociadas.
Financiamiento
Como Parte del Grupo Responsable:
(2021-2023) Subsidio del CONICET: Proyecto de Investigación Plurianual PIP 2021-2023 para el desarrollo del proyecto “Delimitación de especies crípticas en vectores secundarios del Chagas; Estudio de poblaciones de Triatoma garciabesi y Triatoma sordida (Hemiptera: Reduviidae: Triatominae), integrando morfología, reproducción, citogenética y metataxonomía”. Monto total: $1.320.000.
Como Parte del Grupo de Trabajo:
(2023-2026) Subsidio de la Agencia Nacional de Promoción Científica y Tecnológica (ANPCyT): Proyecto de Investigación y Desarrollo en Cannabis, para el desarrollo del proyecto “Daño hepático y Cannabis. Evaluación de los efectos de Aceites de Cannabis sobre un modelo de daño hepático en ratones. Respuesta del sistema redox celular y del Sistema Endocannabinoide”. Monto total: $9.950.000.
(2023-2026) Subsidio de la ANPCyT: Proyecto de Investigación y Desarrollo en Cannabis, para el desarrollo del proyecto “Caracterización de variedades de Cannabis sativa L. empleadas terapéuticamente en nuestra región. Aspectos morfoanatómicos, químicos y marcadores moleculares: herramientas necesarias para el manejo y el fitomejoramiento de cultivares”. Monto total: $9.800.000.
Como Investigador Responsable:
(2015-2019) Subsidio de la Agencia Nacional de Promoción Científica y Tecnológica (ANPCyT) (PICT-2014-3632), obtenido en el marco de la convocatoria PICT-2014 Cooperación Internacional CABBIO, para el desarrollo del Proyecto “Evaluación de RNAi como estrategia de biocontrol para Lutzomyia longipalpis, vector de Leishmaniasis Visceral”. Monto total: $400.000
(2011-2015) Subsidio de la ANPCyT (PICT PRH 112) obtenido en el marco del PRH 36 para el desarrollo del Proyecto “Metagenómica de Microorganismos”. Monto total: $300.000
(2013-2015) Subsidio de la UNNOBA obtenido en el marco de la Convocatoria para la Acreditación de Proyectos y Solicitud de Subsidios de Investigación Bianuales (SIB 2013) (Exp. 2581/2012), para el desarrollo del Proyecto “Análisis metagenómico de microorganismos asociados a suelos de ambientes extremos con potencial para biorremediación”. Monto total: $3.000
(2011-2013) Subsidio del CONICET (PIP 0294) obtenido en el marco de la Convocatoria Proyectos de Investigación Plurianuales Integrante único para el desarrollo del Proyecto “Caracterización Metagenómica de los Microbios Asociados a Lutzomyia longipalpis, Vector de Leishmaniasis Visceral”. Monto total: $36.000 (vencimiento proyecto: 05/11/15)
(2010-2012) Subsidio de la UNNOBA obtenido en el marco de la Convocatoria para la Acreditación de Proyectos y Solicitud de Subsidios de Investigación Bianuales (SIB 2010) (Exp. 1388/2010), para el desarrollo del Proyecto “Caracterización Metagenómica de los Microbios Asociados a Lutzomyia longipalpis, Vector de Leishmaniasis Visceral”. Monto total: $1.000
(2008-2010) Subsidio Fundación Bunge y Born (FBBEI5/08) obtenido por concurso para el desarrollo del Proyecto “Caracterización metagenómica de los microbios asociados a especies de Lutzomyia, vectores de Leishmaniasis”. Monto total: $100.000
(2008-2010) Subsidio de la UNNOBA obtenido en el marco de la Convocatoria 2008 para la Acreditación de Proyectos (Resolución 970/08), para el desarrollo del Proyecto “Caracterización de las comunidades virales del suelo desde una perspectiva agrícola”. Monto total: $1.000
Como Parte del Grupo Responsable:
(2019-2022) Subsidio de la Comisión de Investigaciones Científicas de la Provincia de Buenos Aires (CIC) para Ideas-Proyecto de Investigación en Temas Prioritarios y de Impacto para la Provincia de Buenos Aires, para el desarrollo del proyecto “Transcriptómica aplicada al estudio de las secreciones salivales en hemípteros plagas del maíz (Hemiptera: Delphacidae)” (Resolución Nº 801/18). Monto total: $200.000
Como Investigador Participante:
(2013-2014) Subsidio de la ANPCyT Proyectos de Investigación Científica y Tecnológica Orientados (2011) ANLIS-Categoria II – Equipo de Trabajo (PICTO-2011-0161) para el desarrollo del Proyecto “Eco-epidemiología, biología y distribución de Phlebotominae vectores de las Leishmania sp, en las regiones NOA, NEA y Chaqueña”. Investigador Responsable: María Soledad Santini. Co-Investigador Responsable: Oscar D. Salomón. Monto total: $181.500
(2013-2014) Subsidio de la UNLP obtenido en el marco de la Convocatoria para la Acreditación de Proyectos de Investigación y Desarrollo para el año 2013 (11-X669) para el desarrollo del Proyecto “Análisis y registro de la biodiversidad de la producción hortícola en la región Capital de la provincia de Buenos Aires”. Investigador Responsable: Andrés N. McCarthy. Monto total: $8.500
Publicaciones
Estas son las publicaciones más destacadas realizadas en los últimos años:
- Vaccarini, C., Mercado, M. I., Ponessa, G. I., McCarthy, A., McCarthy, C., Cattáneo, R. A., Sedan, D., & Andrinolo, D. (2025). Morphoanatomical, histochemical, and molecular characterization of female Cannabis plants from three Argentine strains. Protoplasma. https://doi.org/10.1007/s00709-025-02054-0
-
McCarthy, C. B. (2024). Current status of metatranscriptomic and related studies in hematophagous disease-transmitting vectors. Journal of the Florida Mosquito Control Association, 71(1). https://doi.org/10.32473/jfmca.71.1.135288
-
Kenis, M., Benelli, G., Biondi, A., Calatayud, P.-A., Day, R., Desneux, N., Harrison, R. D., Kriticos, D., Rwomushana, I., van den Berg, J., Verheggen, F., Zhang, Y.-J., Agboyi, L. K., Ahissou, R. B., Ba, M. N., Bernal, J., Bueno, A. de F., Carrière, Y., Carvalho, G. A., Chen, X.-X., Cicero, L., du Plessis, H., Early, R., Fallet, P., Fiaboe, K. K. M., Firake, D. M., Goergen, G., Groot, A. T., Guedes, R. N. C., Gupta, A., Hu, G., Huang, F. N., Jaber, L. R., Malo, E. A., McCarthy, C. B., Meagher Jr., R. L., Mohamed, S., Mota Sanchez, D., Nagoshi, R. N., Nègre, N., Niassy, S., Ota, N., Nyamukondiwa, C., Omoto, C., Palli, S. R., Pavela, R., Ramirez-Romero, R., Rojas, J. C., Subramanian, S., Tabashnik, B. E., Tay, W. T., Virla, E. G., Wang, S., Williams, T., Zang, L.-S., Zhang, L., & Wu, K. (2022). Invasiveness, biology, ecology, and management of the fall armyworm, Spodoptera frugiperda. Entomologia Generalis, 43(2), 187–241. https://doi.org/10.1127/entomologia/2022/1659
- Rozadilla, G., Moreiras Clemente, J., & McCarthy, C. B. (2020). HoSeIn: A Workflow for Integrating Various Homology Search Results from Metagenomic and Metatranscriptomic Sequence Datasets. Bio-protocol, 10(14): e3679. https://bio-protocol.org/e3679
- Rozadilla, G., Cabrera, N. A., Virla, E. G., Greco, N., & McCarthy, C. B. (2020). Gut microbiota of Spodoptera frugiperda (J.E. Smith) larvae as revealed by metatranscriptomic analysis. Journal of Applied Entomology, 00, 1–13. https ://doi.org/10.1111/jen.12742
- Caligiuri, L. G., Sandoval, A. E., Miranda, J. C., Pessoa, F. A., Santini, M. S., Salomón, O. D., Secundino, N. F. C., & McCarthy, C. (2019). Optimization of DNA Extraction from Individual Sand Flies for PCR Amplification. Methods and Protocols, 2(2), 36. https://www.mdpi.com/2409-9279/2/2/36
- Caligiuri, L. G., Sandoval, A. E., Miranda, J. C., Pessoa, F. A., Santini, M. S., Salomón, O. D., Secundino, N. F. C., & McCarthy, C. (2018). Optimisation of DNA Extraction from Individual Sand Flies for PCR Amplification. Preprints. DOI: 10.20944/preprints201812.0189.v1.
- McCarthy, C., Rozadilla, G., & Moreiras Clemente, J. (2018). HoSeIn: A Workflow for Integrating Various Homology Search Results from a High-Throughput Sequence Dataset. Preprints, https://www.preprints.org/manuscript/201812.0189/v1
- McCarthy, C., Carrea, A., & Diambra, L. (2017). Bicodon bias can determine the role of synonymous SNPs in human diseases. BMC Genomics, 18, 227. https://doi.org/10.1186/s12864-017-3609-6
- McCarthy, C. B., & Colman, D. I. (2015). Soil metagenomes from different pristine environments of Northwest Argentina. Genome Announcements, 3(4): e00926-15. https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-017-3609-6
- McCarthy, C. B., Cabrera, N. A., & Virla, E. G. (2015). Metatranscriptomic analysis of larvae guts from field-collected and laboratory-reared Spodoptera frugiperda from the South American subtropical region. Genome Announcements, 3(4): e00777-15. https://mra.asm.org/content/3/4/e00777-15
- Haase, S., McCarthy, C., Ferrelli, M. L., Pidre, M. L., Sciocco-Cap, A., & Romanowski, V. (2015). Development of a recombination system for the generation of occlusion positive genetically modified Anticarsia gemmatalis Multiple Nucleopolyhedrovirus. Viruses, 7, 1599–1612. https://www.mdpi.com/1999-4915/7/4/1599
- Caligiuri, L. G., Acardi, S. A., Santini, M. S., Salomón, O. D., & McCarthy, C. B. (2014). Polymerase chain reaction (PCR)-based assay for the detection and identification of sand fly gregarines in Lutzomyia longipalpis, vector of visceral leishmaniasis. Journal of Vector Ecology, 39(1), 83–93. https://onlinelibrary.wiley.com/doi/full/10.1111/j.1948-7134.2014.12074.x
- Monteoliva, D., McCarthy, C. B., & Diambra, L. (2013). Noise Minimisation in Gene Expression Switches. PLOS ONE, 8(12): e84020. https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0084020
- McCarthy, C. B., Santini, M. S., Pimenta, P. F. P., & Diambra, L. A. (2013). First comparative transcriptomic analysis of wild adult male and female Lutzomyia longipalpis, vector of Visceral Leishmaniasis. PLOS ONE, 8(3): e58645. https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0058645
- Polverino, D., Mariñelarena, A. J., McCarthy, C. B., & Rivera Pomar, R. V. (2012). Culture-independent characterisation of the bacterioplankton community composition of a mesotrophic reservoir (Embalse Río III, Argentina). Revista Mexicana de Biodiversidad, 83, 548–552. http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S1870-34532012000200026
- McCarthy, C. B., Diambra, L. A., & Rivera Pomar, R. V. (2011). Metagenomic analysis of taxa associated with Lutzomyia longipalpis, vector of visceral leishmaniasis, using an unbiased high-throughput approach. PLOS Neglected Tropical Diseases, 5(9): e1304. https://journals.plos.org/plosntds/article?id=10.1371/journal.pntd.0001304